免疫组织化学检测(IHC)是病理诊断中常用的一种检测手段,它是利用带标记的特异性抗体(或抗原)与组织内抗原(或抗体)进行特异性结合并通过化学反应使标记抗体显色,以此来对组织细胞内抗原进行定位、定性及定量的研究。
PD-L1免疫组化检测,作为当前肿瘤治疗领域中最具前景的研究方向之一,肿瘤免疫治疗是通过调动机体的免疫系统,增强抗肿瘤免疫力,从而抑制和杀伤肿瘤细胞
PD-L1免疫组化检测是预测PD-1/PD-L1抑制剂疗效的一种简单有效的方法,目前,已被FDA/NMPA批准的PD-1/PD-L1抑制剂的适应症、PD-L1检测平台和判读方法,可参考:总结:PD-L1免疫组化检测难点与要点
免疫组织化学抗体试剂及检测试剂是指一类利用免疫学原理结合酶催化底物显色的化学方法,检测组织标本中抗原的检测试剂。此类试剂为待测抗原特异性单克隆或多克隆抗体,或抗体与显色系统、对照试剂、质控片及其它辅助试剂一同包装成试剂盒形式的检测试剂盒。
我们查看PD-L1的一些临床研究报告,如:PD-L1 IHC 22C3 pharmDx 中可看出,评估PD-L1免疫组化检测试剂盒/抗体的performance:
用一批试剂盒分别进行进行仪器间和批内、操作员间、日间、同一轮检测内、日 内的检测。采用 bootstrap 法计算平均阴性百分比一致率 (ANA)、平均阳性百分比一致率(APA)和总体一致率(OA), 双侧 95%置信区间。对于一致性结果为 100%的研究,采用Wilson Score 法计算 TPS ≥ 1%和 TPS ≥ 50%临界值的阴性百分比一致率(NPA)、阳性百分比一致率(PPA)和总体一致率(OA),双侧 95%置信区间。
Calculation
阳性一致性(positive percent agreement, PPA)和阴性一致性(negative percent agreement, NPA)比较好理解,但是在Immunological Precision研究中,主要是想看两个paired readers之间评估结果的一致性,而PPA和NPA在此情况下并没有假设哪个reader作为reference(这个reference是在PPA/NPA中需要定义的),所以当选不同reader作为reference时,两个结果会不一样;因此在CLSI-I/LA28-A2中提到,采用平均阴性百分比一致率 (average negative agreement, ANA)、平均阳性百分比一致率(average positive agreement, APA) 即weighted average的NPA/PPA。
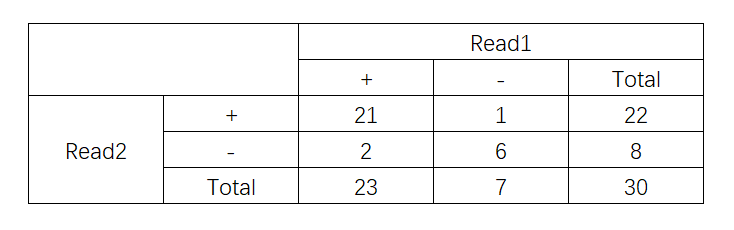
以上表数据,APA和ANA计算公式如下:
APA = 21/23*23/(23+22) + 21/22*22/(23+22),即APA = 2*21/(23+22) = 0.933ANA = 6/7*7/(7+8) + 6/8*8/(7+8),即ANA = 2*6/(7+8) = 0.8
Confidence Interval
CLSI-I/LA28-A2中建议采用percentile bootstrap方法,理由是:方便简单并且足够解释APA/ANA的置信水平;从一些其他的资料来看,也会采用Transformed Wilson方法
Bootstrap方法是一类非参数Monte Carlo方法,其通过再抽样对总体分布进行估计,再抽样方法将观测到的yy样本视为一个有限的总体,从中进行随机(再)抽样来估计总体的特征以及对抽样总体做出统计推断,当目标总体分布没有指定时,Bootstrap方法经常被使用,此时样本是唯一已有的信息
对于“再抽样”来说,Jackknife也是一个再抽样的方法,但是其更加类似于“leave-one-out”的交叉验证的方法;其在估计值不光滑的情况下,可能会失效(如中位数);对于线性统计量的估计方差这个问题,Jackknife或者Bootstrap会得到同样的结果,但在非线性统计量的方差估计问题上,刀切法严重依赖于统计量线性的拟合程度,所以远不如自助法有效。(部分参考自参考自:https://www.jianshu.com/p/7ce411e3c377)
常见的Bootstrap计算渐近置信区间的方法有:
- 标准正态Bootstrap置信区间
- 基本Bootstrap置信区间
- Bootstrap百分位数(percentile)置信区间
- Bootstrap t置信区间
其中percentile bootstrap方法是重复样本的估计值分布中,取百分位数来估计置信区间,一般需要找到分布中 10% 分位和 90% 分位数对应估计值,从而构建成置信区间;这里的假设是重抽样构建的估计值分布是总体分布的一个很好的近似
bootstrap计算方式可直接用boot包的boot和boot.ci函数,以上述数据计算APA作为例子,如计算95%的置信区间:
library(boot)
set.seed(123)
x <- runif(100)
testmedian <- function(x, d) {
return(median(x[d]))
}
b <- boot(x, testmedian, R=1000)
bci<- boot.ci(b, conf = 0.95, type = c("perc"), index = 1)
print(bci$percent[, c(4,5)])其中boot.ci的步骤也可以使用quantile函数来代替(type指定分位数算法),如:
quantile(b$t, probs = c(0.025, 0.975))上述Bootstrape内容参考自:
- Lecture 7: Bootstrap(自助)方法和Jackknife(刀切)方法
- Understanding Bootstrap Confidence Interval Output from the R boot Package
至于Transformed Wilson方法,其实是指将APA/ANA根据公式转化一下,使得其符合Wilson的方法,如下所示:
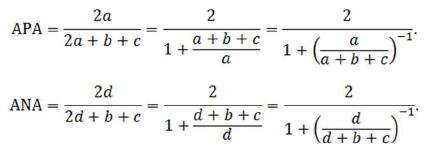
那么以计算APA的CI为例,先用a/(a+b+c)计算其wilson的置信区间,然后通过(2*lower/(1+lower), 2*upper/(1+upper))转化得到APA对应的置信区间,如:
res <- DescTools::BinomCI(a, a+b+c, method = "wilson")
ci <- c(2 * res[1,2] / (1 + res[1,2]), 2 * res[1,3] / (1 + res[1,3]))本文出自于http://www.bioinfo-scrounger.com转载请注明出处