在之前的一篇博文Alignment-free的转录本比对工具-Salmon提到了用Alignment-free的Salmon来基于转录本水平进行表达丰度的定量。 我最早接触转录本的定量则是Alignment-based transcript quantification,也就是先比对后定量。我最先是在无参转录组中用了RSEM,因为无参转录组需要对reads进行拼接,然后将reads比对至拼接的转录本上,再通过定量获得其表达丰度。由于那时Trinity软件的分析流程推荐定量软件中有RSEM(当然不止推荐了一种,基于比对的还有eXpress,但那时网上可搜到的教程几乎都推荐使用RSEM),所以我就挑了RSEM来做定量分析。但只是按照Trinity分析流程的代码来使用RSEM(因为Trinity将RSEM的使用包装在perl代码里面了,包括使用步骤以及参数),那时只能说是很粗浅的接触了下。所以这次我准备从头好好理解下RSEM这个转录本定量软件
Download and install
RSEM是在2010年发表的,最新更新是在2016年,下载地址则http://deweylab.github.io/RSEM/,下载后编译下即可
make
make installRSEM整体上来说是属于定量软件,但其支持调用其他比对软件,如Bowtie,Bowtie2和STAR,来将reads比对至转录本上,所以必须至少预先安装上述3款比对软件中的一种(如果没安装,可以直接在RSEM安装目录下编译一个bowtie2,因为其在RSEM软件安装包中附带)
Build References
这步可以理解为是对转录本建索引,RSEM支持两种方式:
第一种是提供参考基因组fa序列和GTF注释文件,那么RSEM则会通过GTF注释文件从参考基因组序列中提取出各个转录本的序列,然后利用Bowtie2 or STAR等软件来建索引
~/biosoft/rsem/RSEM-1.3.0/rsem-prepare-reference -gtf ~/annotation/hg38/gencode.v27.annotation.gtf --bowtie2 ~/reference/genome/hg38/GRCh38.p10.genome.fa ~/reference/index/RSEM/hg38/hg38这里用bowtie2做为测试数据的refences索引,一方面是因为用STAR的话对内存要求较高;另一方面是因此我的测试数据是100bp的双端RNA-SEQ(同Alignment-free的转录本比对工具-Salmon),bowtie2相比bowtie更加适合用于比对
第二种则直接提供转录本序列,然后再建索引(无参转录组一般是这样的);如果还想有基于基因水平的定量结果,则需再加
--transcript-to-gene-map参数,用于导入转录本和基因的对应关系的文件(一列基因ID,一列对应的转录本ID)
在~/reference/index/RSEM/hg38/目录下包含有从参考基因组提取出来的转录本的序列文件()以及bowtie2的索引文件(以.bt2结尾)等
Calculate expression
用RSEM的rsem-calculate-expression命令来对reads进行bowtie2比对以及表达水平的定量
for i in $(seq 49 52);do ~/biosoft/rsem/RSEM-1.3.0/rsem-calculate-expression \
--paired-end -p 6 --bowtie2 --append-names --output-genome-bam \
SRR62690${i}.clean_1.fastq SRR62690${i}.clean_2.fastq \
~/reference/index/RSEM/hg38/ RSEM/SRR62690${i};done--paired-end 表明是双端测序数据
--bowtie2 指定bowtie2来用于reads比对
--append-names 用于在结果中附加上gene name和transcript name
--output-genome-bam 结果中输出基于基因水平的bam文件(默认只有转录水平的bam文件)
--estimate-rspd 文档中的例子还加了这个参数,用于estimate the read start position distribution,看是否有positional biases
RSEM调用bowtie2的参数可以通过--bowtie2-mismatch-rate,--bowtie2-k以及--bowtie2-sensitivity-level来设定,而默认的参数如下:
bowtie2 -q --phred33 --sensitive --dpad 0 --gbar 99999999 \
--mp 1,1 --np 1 --score-min L,0,-0.1 -I 1 -X 1000 --no-mixed \
--no-discordant -p 6 -k 200 -x /home/anlan/reference/index/RSEM/hg38/ \
-1 SRR6269049.clean_1.fastq -2 SRR6269049.clean_2.fastq | samtools view -S -b -o RSEM/SRR6269049.temp/SRR6269049.bam从上述--gbar可看出,RSEM是不支持有gap的比对;--no-mixed则表示RSEM不支持单个reads的比对;--no-discordant则告诉我们RSEM也不支持paired reads discordant的比对;-k 200表明RSEM希望bowtie2输出最佳的200个比对结果
所以如果想自己设定bowtie2的比对参数,则必须考虑以上几点要求,RSEM可不支持默认参数的比对结果(也就是说,会报错!)。在保证上述情况下,可先自行使用bowtie2其他任意比对参数,然后再将比对后的bam文件作为输入RSEM定量的输入文件即可
Output
先看下输出的结果文件有哪些:
每个样本有两个bam文件,这是因为有--output-genome-bam参数的;而且也可以看到两个bam文件比一般转录组比对后的bam文件都要大好多,这是因为RSEM调用bowtie2软件进行比对时,用了-k 200参数,所以RSEM跟其他的一些软件不一样,其是考虑多重比对的reads的,而不是像Salmon只考虑最佳比对情况?
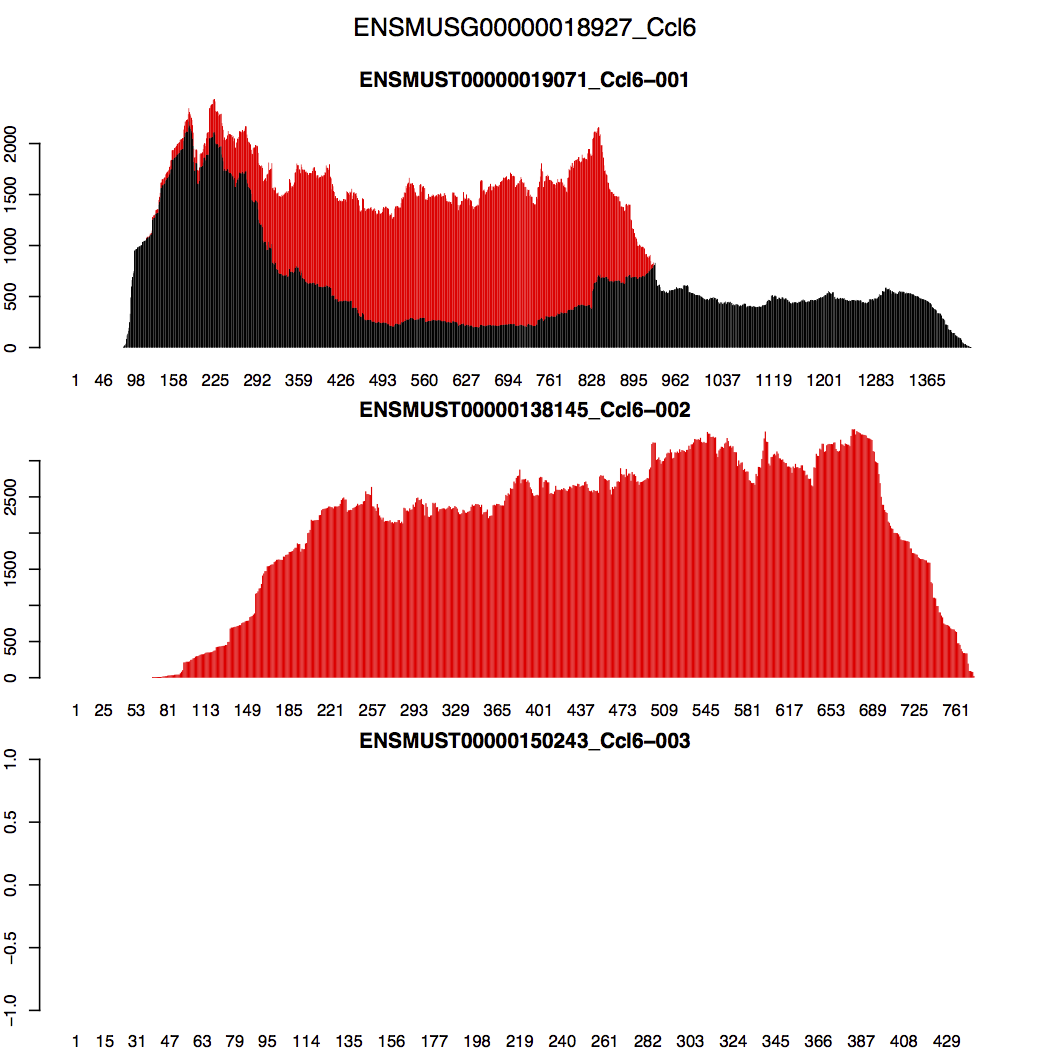
对于RSEM如何处理multimapping reads,RSEM官方教程特意讲了一点,以Ccl6基因的3个转录本的比对结果为例,其第2个转录本全长与第1个转录本重叠,所以在这重叠区域会产生大量的multimapping reads。RSEM的处理方法是这样的:RSEM先尽量让第1个转录本在其重叠区域的reads(包括unique reads和multimapping reads)分布趋于平滑?因此再将在区域的剩下multimapping reads算在第2个转录本上;至于第3个转录本,其没有reads比对上,所以就空的;总体上如下图所示(来自于官网教程)
剩下的最后两个文件.genes.results和.isoforms.results则分别是基于基因水平和转录水平的定量结果,以.isoforms.results为例
- 第1,2列分别是transcript id和gene id,如果像我一样设置了
--append-names参数,那么在ID后面还会附上transcript name和gene name(以下划线_分隔)- 第3,4列分别是transcript length和effective_length,这里是length是没有包括转录本的poly(A) tail的,而effective_length则是指可以有fragment覆盖的区域,计算公式:transcript length - mean fragment length + 1;如果是
.genes.results中的length,则是所有transcript length的加权平均数- 第4,5,6列则分别是expected_count,TPM,FPKM;expected_count比较复杂,跟一般的count数定义有点不一样,是指the sum of the posterior probability of each read comes from this transcript over all reads,RSEM给出的解释是(复制黏贴了):Because 1) each read aligning to this transcript has a probability of being generated from background noise; 2) RSEM may filter some alignable low quality reads, the sum of expected counts for all transcript are generally less than the total number of reads aligned;TPM和FPKM则比较常见了
- 第7列则是IsoPct,其含义是转录本上的丰度占其基因上的丰度的百分比
最后应该是根据上述定量的expected_count进行差异表达分析,一般会选择最为常用的DESeq2和EdgeR等差异表达分析软件,而RSEM则建议使用EBSeq,因为RSEM认为前者两款软件并没有将read mapping的不确定性所导致的变异所考虑进去(个人认为是不是就是multimapping那部分reads?),而EBSeq则充分考虑该情况。。。反正我也还是不懂其原理。。。
除了上述的内容,RSEM还对结果进行了可视化,具体可参照https://github.com/deweylab/RSEM
PS. RSEM还支持chip-seq数据和scRNA-seq数据。。。下次有机会再看看
本文出自于http://www.bioinfo-scrounger.com转载请注明出处