之前学习了ChAMP包来处理甲基化芯片分析的整个常规流程,这个包整合了好多常用工具以及分析算法,对使用者来说非常的便捷;但是从其说明文档来看,对于一些比较基础的过程讲的比较少,作为主要的读入芯片数据那步来说,我还是没明白芯片数据是怎么转化为beta矩阵的,所以我找了minfi包来了解下这个过程。 ChAMP包的读入数据也是利用minfi包的方法,所以两者在这个点上是一致的。minfi包的分析流程结构如下(取自minfi文档):
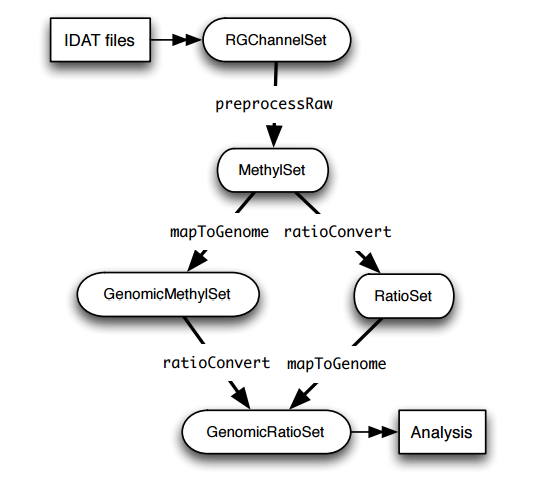
从IDAT files到GenomicRatioSet这个可用于下游分析(差异分析)的对象,主要经过RGChannelSet(probe level),MethylSet(CpG locus level),GenomicMethylSet(mapped to genome) or RatioSet(Beta and/or M (logratio of Beta))这些步骤
如果只是简单的使用minfi包,那么步骤如下:
读入IDAT文件,也想
ChAMP包一样准备好一个sample_sheet.csv的文件,放在R的当前目录下(该目录下只能有其一个csv文件)library(minfi) targets <- read.metharray.sheet("./") RGset <- read.metharray.exp(targets = targets) pd <- pData(RGset) annotation(RGset) #查看所会用到的注释文件有哪些如果没有
sample_sheet.csv文件也是可以读入IDAT数据的,只是会没有一些样品信息对读入后的原始数据进行预处理,bioconductor的minfi处理甲基化数据这篇文章大概列了几种预处理的函数,这里就用Illumina的标准处理(类似于 performed by Genome Studio)
pRGset <- preprocessIllumina(RGset) mRGset <- mapToGenome(pRGset)除了用这个外,还可以自己定义过滤标准,类似于
ChAMP包封装的那些标准,自己用R代码来过滤,比如https://github.com/wkl1990/illumina-450K-analysis/blob/master/R/450K_pipeline.R,这样好处是直观并且了解了过滤过程还有一些标准化函数,如:
preprocessSWAN,preprocessQuantile以及preprocessFunnorm,(看着眼熟,原来都整合在ChAMP包里了)接着转化为
GenomicRatioSet对象dat <- ratioConvert(mRGset,type="Illumina")最后设置个分组矩阵,然后就是差异甲基化位点分析,比如用下
limma包library(limma) bate <- getBeta(dat) design <-model.matrix(~pd$Sample_Group) fit <- lmFit(bate, design) fit2 <- eBayes(fit, trend=TRUE) output <- topTable(fit2, coef=2,n=Inf)当然minfi中差异分析则不是用limma的,而是
dmpFinder函数,下面是文档的例子dmp <- dmpFinder(mset, pheno=pd$Sample_Group, type="categorical")如果是Differential Methylation Regions分析,则使用
bumphunter函数library(doParallel) detectCores() registerDoParallel(cores = 6) design <-model.matrix(~pd$Sample_Group) res <- bumphunter(dat, design, cutoff=0.1)
结合上述的过程,如果单纯的简单的分析下差异,minfi包也是可以毫无问题的,如果还需要进行一点质控以及可视化,则需要自行写脚本了;所以如果想方便,那么ChAMP包则是更好的选择吧,过滤啥的都一并处理了,可真省事了。。。
最后推荐一篇博文甲基化特异性区域的计算鉴别,该作者我甚是佩服,进行了多个组学文章分析过程的重现,而且也附上代码,是一个很好的实践教程,这篇则是多形性成胶质细胞瘤(GBM)甲基化分析的文章。
还有一篇也是在上述文章上看到的,bioconductor中甲基化芯片的分析流程: A cross-package Bioconductor workflow for analysing methylation array data,也是一个很好的实践教程,我也打算学习下(最主要主要提供了原始数据!!!)
本文出自于http://www.bioinfo-scrounger.com转载请注明出处