之前在生信媛公众看到一篇软文GOplot 可视化基因富集分析结果,刚好我也对一些可视化方法蛮感兴趣,因此学习了下,感觉蛮有意思的,其提出了一些新的GO富集可视化思路;因此在官方提供的测试数据基础上,尝试用下常规的GO富集数据格式
GOplot使用了zscore概念,其并不是指Z-score标准化,而是用于表示参与某个GO Term下基因的上下调情况,公式:
\[ zscore=\frac{(up-down)}{\sqrt{count}} \]
zscore is an easy to calculate value to give you a hint if the biological process (/molecular function/cellular components) is more likely to be decreased (negative value) or increased (positive value)
GOplot使用说明可以参照官方文档:https://wencke.github.io/,其对于这个R包的使用讲的非常的详细,看一遍就懂了
文档使用的是EC数据集,但实际使用中则是各个富集软件来源的结果,因此我们需要对于自己的数据先进行预处理,以满足GOplot调用规则;其有circle_dat函数可以用于整合外来数据,但是要求数据的格式(尤其指列名)规范化,我们可看函数说明或者EC示例文件,模仿着修改
比如我整理的两个文件:DEG 和 enrichment
导入数据生成规范格式
library(GOplot)
deg <- read.table(file = "../Desktop/DEG.txt", sep = "\t", header = T, stringsAsFactors = F)
enrich <- read.table(file = "../Desktop/enrichment.txt", sep = "\t", header = T, stringsAsFactors = F)
circ <- circle_dat(enrich, deg)接着看下GOplot所能绘制的几类图片,个人觉得可视化展示方法的新意才是值得学习的,至于用什么软件展示则是次要的;当然单纯从美观角度来说,GOplot出具的图片还是可以的
首先是barplot图,纵坐标是adj.pvalue,横纵标是GO id,通过柱子颜色来展示zscore
GOBar(circ, display = 'multiple')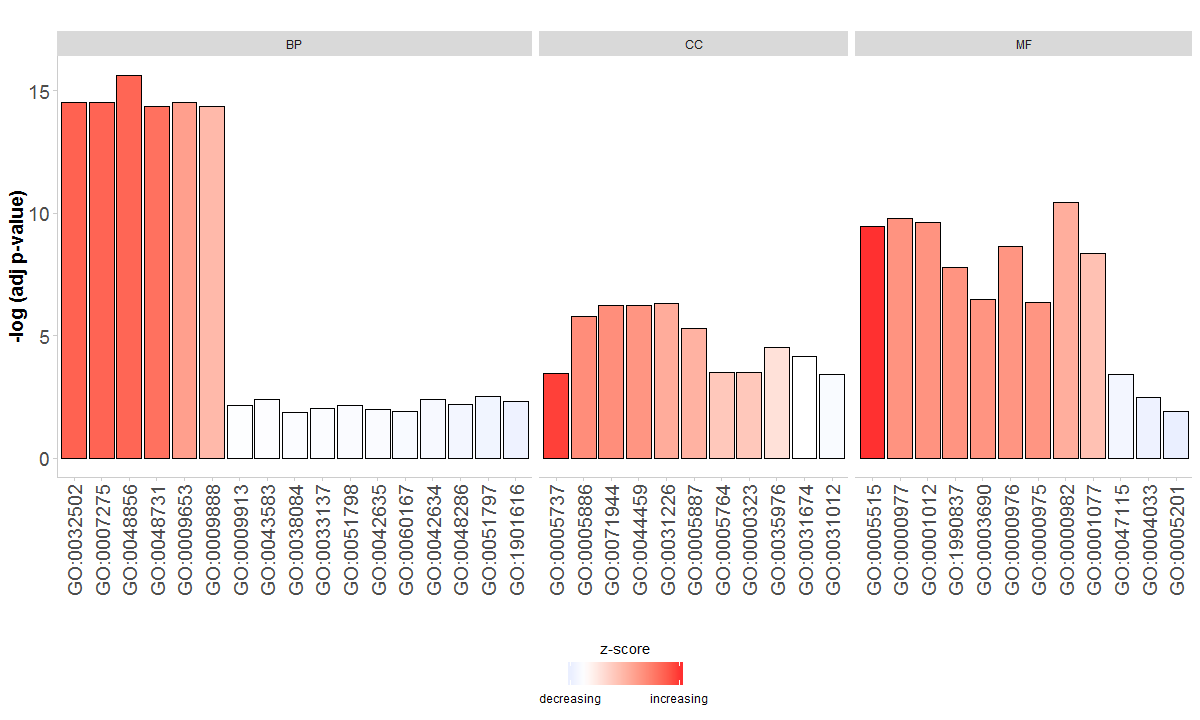
bubble图,以adj.pvalue为纵坐标,zscore为横纵标,对于满足一定阈值(比如log10(adj.pvalue) > 3)的显示GO id(PS.这里的纵坐标写着是log是指log10)
GOBubble(circ, display = "single", labels = 3, table.legend = F)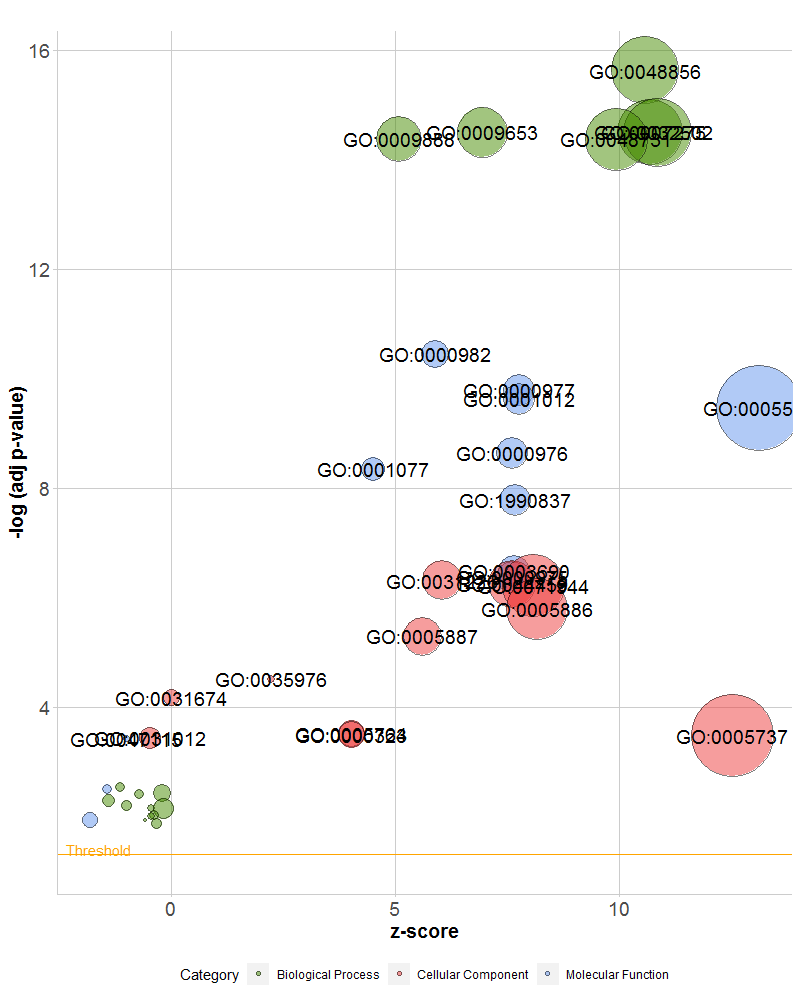
GOplot为了过滤一些比较相似的GO ID(认为是冗余的),其根据不同GO之间参与基因的相似程度进行的过滤,这个可以自己写代码预先过滤下,也可以直接用其写好的函数reduce_overlap
reduced_circ <- reduce_overlap(circ, overlap = 0.75)
GOBubble(reduced_circ, labels = 3, table.legend = F)然后是Circular图,相当有圈圈图,内圈展示zscore,外圈展示GO id,外圈里面则是参与基因的上下调分布情况
GOCircle(circ, nsub = 10, label.size = 5, rad1 = 3, rad2 = 4, table.legend = F)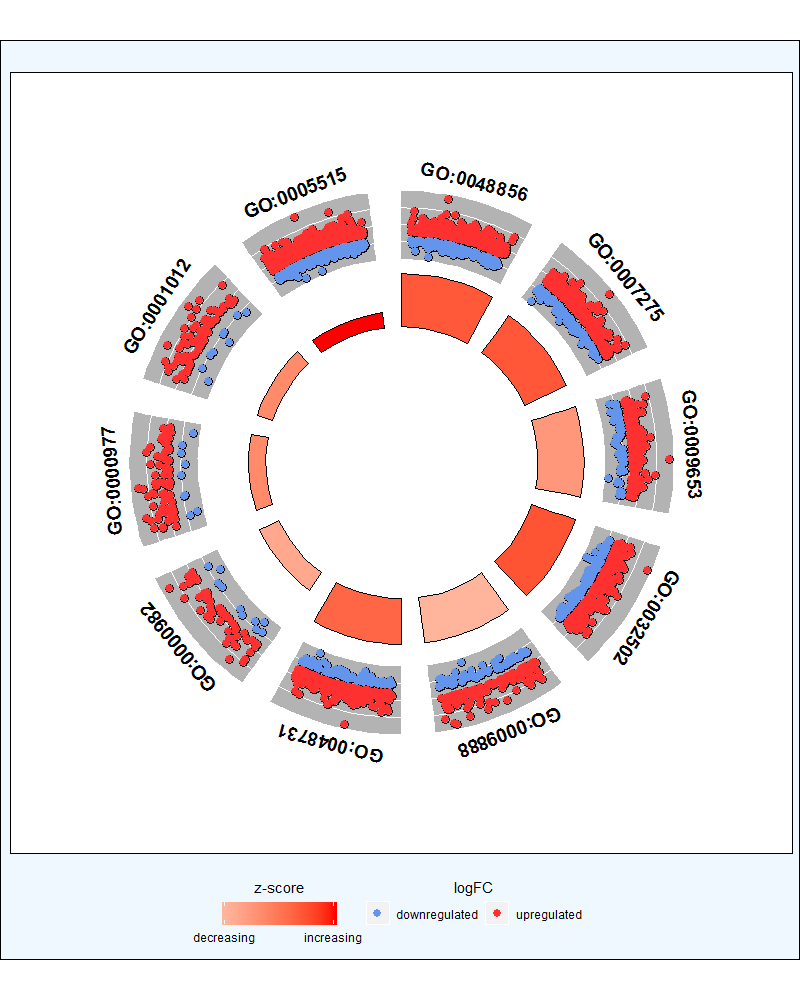
还有则是用圈圈图展示gene和go term之间的关系,这需要先用chord_dat函数构建一个对象,其需要circ,deg以及制定的go id等数据;limit参数则是限制基因/GO的数目
process <- tail(enrich$ID, 10)
chord <- chord_dat(data = circ, genes = deg, process = process)
GOChord(chord, space = 0.02, gene.order = 'logFC', gene.space = 0.25, gene.size = 2.5, limit = c(0,2))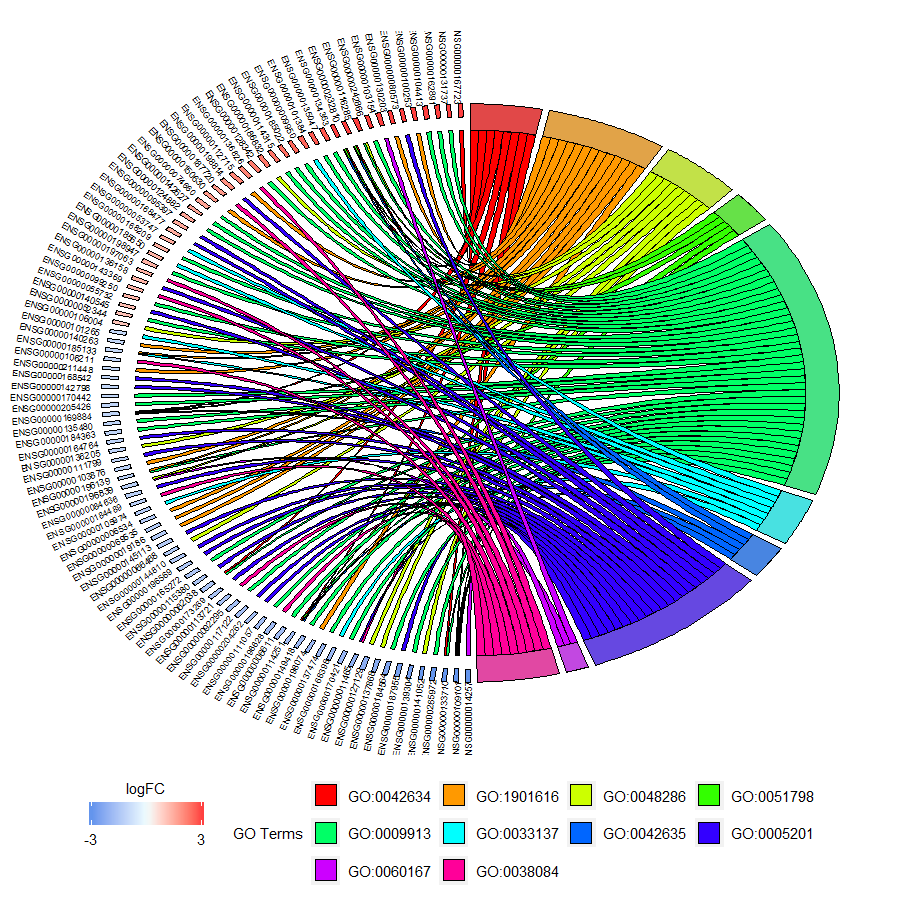
接着则是其热图,这张图比较有意思,其以热图展示GO term和gene id,每行是一个GO,每列是一个gene;如果其图例是count的话,其每格的颜色则是该gene参与图中所有go term总数;如果是logFC的话,则比较好理解,是该基因的logFC的值
GOHeat(chord, nlfc = 1, fill.col = c('red', 'yellow', 'green'))
最后还是一个圈圈图,感觉GOplot把圈圈图的一些展现形式都用了个遍,其内圈是根据基因logFCz做的聚类,外圈则是GO term的堆积图
GOCluster(circ, process, clust.by = 'logFC', term.width = 2)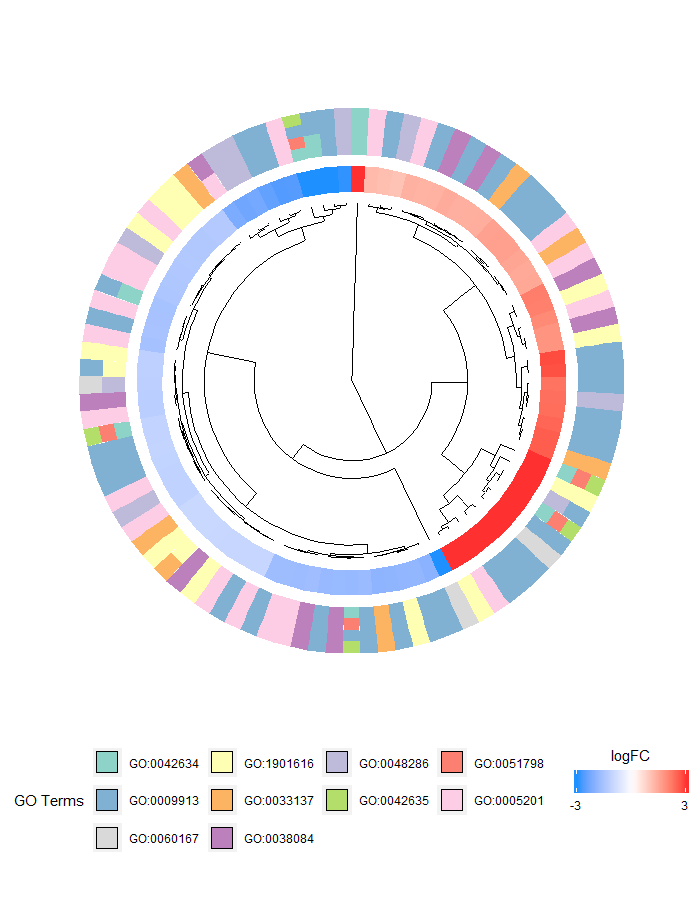
GOplot这个R包的一些展示形式值得我们学习(因为有些绘图细节GOplot处理的不是太好,可选择参数有限),而且可以用于其他方向,不局限于GO富集;这个R包也发表了不错的文章
本文出自于http://www.bioinfo-scrounger.com转载请注明出处