生物信息分析平台可以有效的将非生信人员从生信软件中脱离,只专注于数据的输入与输出,不关注数据的具体分析过程
最近看到一篇可视化生信分析利器 Galaxy:本地部署文章,我也比较喜欢shiny这种工具(可以通过网站进行数据分析以及可视化),这点与Galaxy比较类似,但从平台角度来说,后者更加专业一点 > Galaxy提供了一个执行交互式分析的分析环境,同时确保结果分析具有透明和可重复性。Galaxy框架在其后台封装了很多高端的计算工具,并且在网页的前端给这些工具以直观的使用界面而把计算和存储管理的细节隐藏起来。因此,它消除了在执行许多常见类型的大规模分析过程中对专门的信息学专业知识的需求。正因为如此,使得Galaxy生物信息分析平台成为了目前生物医学研究领域最受欢迎的在线生物信息分析工具。
从上述可看出Galaxy是一个开源平台,然后通过生信分析人员不断以其为基础拓展分析流程,实现多种生信分析工具的融合
Galaxy已整合了多个分析流程的开源工具,通过其网站即可访问使用,进入https://galaxyproject.org/use/,在UseGalxy页面下是官方服务器,点击The Galaxy Project free public server; biomedical research的Server,即可进入免费公共的Galaxy分析平台,我选择NGS:RNA Analysis进入HISAT2分析界面,如下所示:
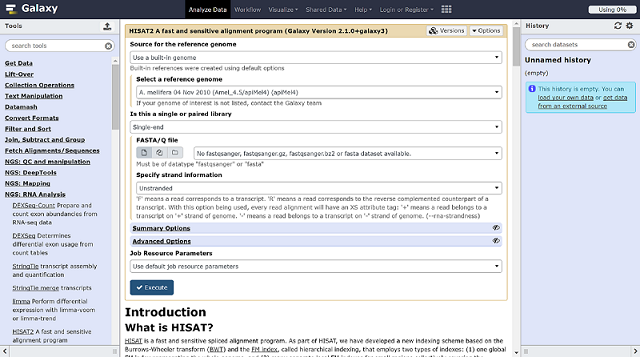
除了流行的分析软件,还有一些文本处理工具(勉强适合一些常规需要,毕竟大家的需求都是多样化的。。。)
除了官方的Galaxy Server外,还有一些团队/个人基于Galaxy开发的公开分析平台,可以在上述网站的Public Servers界面下找到,可以通过一些关键词找到你所需要的分析流程,如搜索proteomics,如下所示:
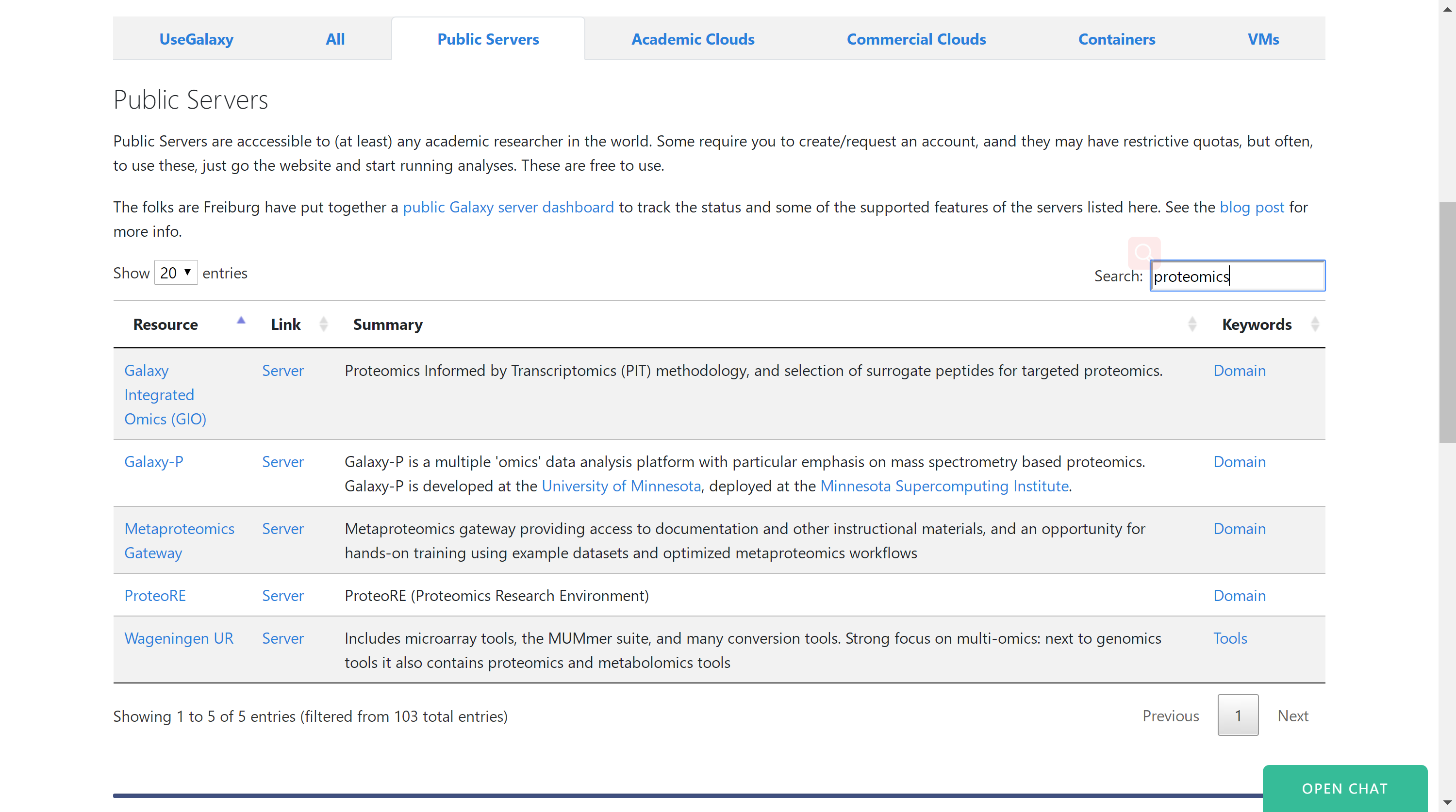
从中可看出Galaxy可以用于搭建定制化的生信分析平台,这点就很有意思了,其主要是提供一个平台框架,而管理员则是需要在这个平台上添加分析工具即可供他人使用,主要还是开源的。。。
参考官方文档Get Galaxy,结合开头那篇博文,可以在linux系统上搭建一个属于自己的生信分析平台了
安装前先检查下是否有Python 2.7,一般ubuntu系统都是默认自带的
先从Git上将最新版Galaxy克隆到本地(现在最新是18.09)
git clone -b release_18.09 https://github.com/galaxyproject/galaxy.git然后运行,该命令第一次运行会安装一些配套文件以及一些Python模块,如果有报错,一般就是缺少一些系统文件,缺啥补啥即可
sh run.sh如果没有报错运行完后,说明Galaxy server已经正常在服务器上运行了,如果想退出的话Ctrl+C即可(如果是将Galaxy提交后台运行,则可以通过kill PID来关闭程序);但是我们不只是希望能在本机登录Galaxy平台(前提你本机上有浏览器),而是想在外部远程登录或者内网下其他电脑登录这个IP,这是需要对Galaxy的配置文件做点修改
首先将初始的配置文件config/galaxy.yml.sample创建出一个新的可使用配置文件config/galaxy.yml(之后Galaxy就识别这个文件中的配置了)
cp config/galaxy.yml.sample config/galaxy.yml然后将默认的本地IPhttp: 127.0.0.1:8080修改为http: 0.0.0.0:8080,8080即端口,这样就允许外部用户访问本机的Galaxy server了(这个跟shiny有点类似),在远程电脑浏览器中输入网址:http://IP:8080/
界面如下所示:
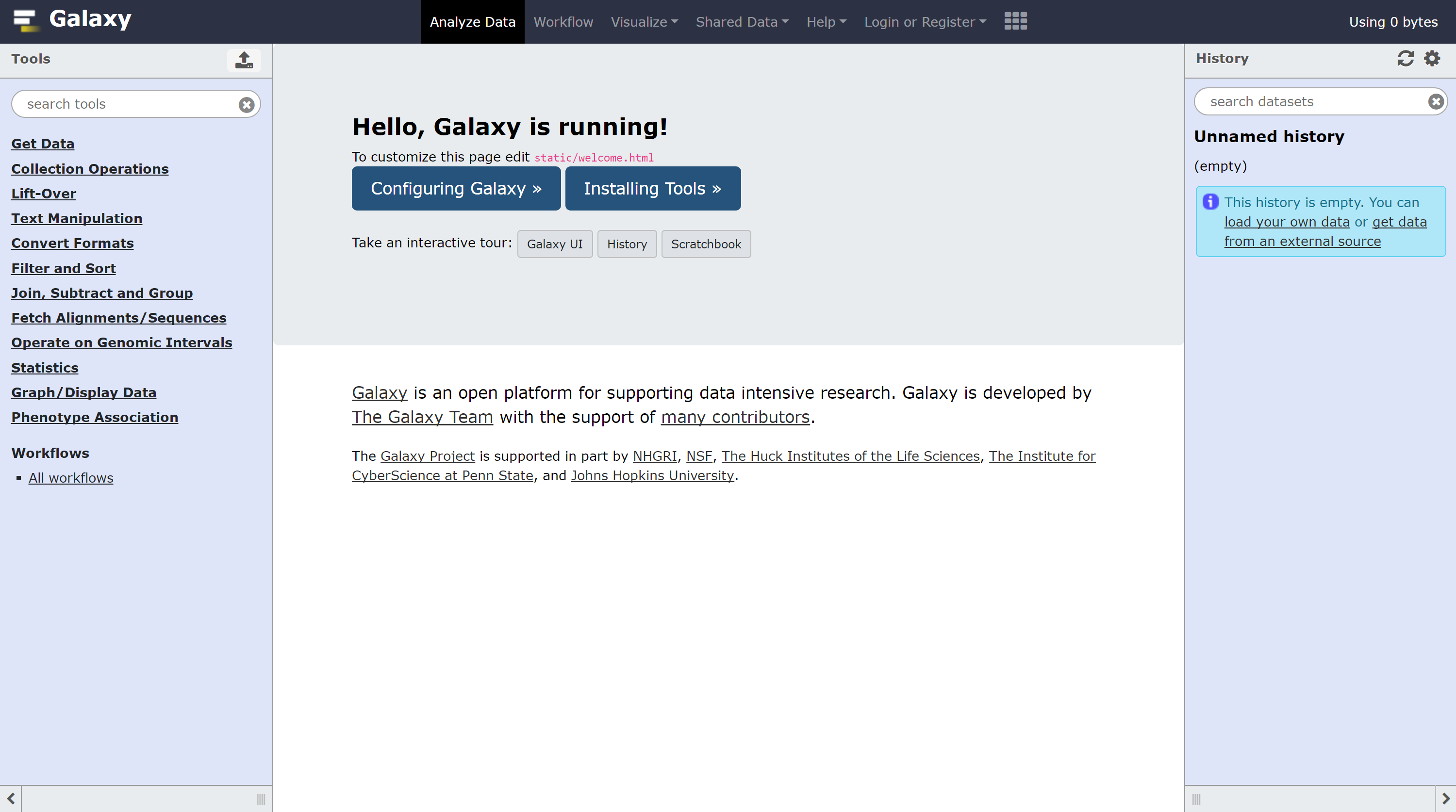
如需安装一些除了自带的一个工具外,可以参考Installing Tools into Galaxy
其实我更好奇如何将自己写的脚本/工具在Galaxy中配置上去,在后续尝试后再整理下
本文出自于http://www.bioinfo-scrounger.com转载请注明出处