之前大致了解了些免疫疗法的相关内容,最近一篇文章深度长文:一文尽览PD-1/PD-L1/CTLA-4肿瘤免疫治疗分子标志物大全概括性的讲述了一些现在免疫疗法中生物标志物的相关内容,值得仔细看一看 先总结下其中一些感兴趣的内容:
最近几年PD-1/PD-L1在一些肿瘤免疫治疗中的突破性的进展令人鼓舞,这也推进了人们对于免疫治疗相关的生物标志物的研究:如常见生化反应手段的PD-L1表达,肿瘤浸润淋巴细胞(TIL),还有听的比较多的微卫星不稳定性(MSI)和错配修复缺陷(dMMR),以及最近研究火热的肿瘤突变负荷(TMB)
从测序的角度来说,可以研究分析的生物标志物(上述提到的)有MSI/dMMR/TMB
近几年不断研究报道TMB与PD-1/PD-L1抑制剂的疗效高度相关,而以TMB为标志物的临床研究大多数都顺利达到终点,几乎没有失败过。这让一些肿瘤患者可以通过TMB标志物还对免疫疗法的疗效进行一定程度的预测
TMB(Tumor mutation burden)既然这么热,那么我需要知道TMB是如何计算的: TMB的定义通常是基因组中每1Mb蛋白编码区的平均突变个数。所以我们可以通过WES全外显子测序,通过call somatic variant(去除胚系DNA变异),然后除以人类蛋白编码区长度MB即可(我觉得。。。)
这篇文章刚好也提到了之前我一朋友推荐我看的一篇文献:Blood-based tumor mutational burden as a predictor of clinical benefit in non-small-cell lung cancer patients treated with atezolizumab,其中提到了bTMB(blood TMB)这个概念,也就是不检测常规的肿瘤组织样的tTMB,而是血液样本的bTMB,从而避免组织样本的一些局限性
文献中bTMB用的筛选指标是:SNVs with ≥0.5% frequency were analyzed, and germline variants and oncogenic driver mutations were removed
而文中所说常规的组织样本的筛选标准:tTMB was defined as the number of somatic, coding, SNVs and indels detected at an allele frequency of ≥5% after the removal of known and probably oncogenic driver events and germline SNPs, as previously described
要想获得靠谱的bTMB,在测序中一定要使用双端样本index (即UDI,针对Illumina平台),否则你将无法排除Index Misassignment带来的样本间污染导致的大量假阳性超低频突变。同时,还需要使用UMI分子标签方法,以排除PCR, 和测序过程中带来的大量错误
说了一些基础概念后,那么怎么计算TMB呢;如果你有突变分析(可以是WES的,也可以是大Panel的)后的MAF格式的文件,那么只需要按照文献中所说的,先过滤掉一些突变位点,然后统计数目再除以蛋白编码区域的长度即可
那么什么是MAF(Mutation Annotation Format ),可以看下这个链接https://docs.gdc.cancer.gov/Data/File_Formats/MAF_Format/,里面除了说了什么MAF外,还对MAF格式的每个列名做了解释说明,一般来说我们从TCGA下载是突变文件都是MAF格式的
如果是VCF格式的突变分析结果,那么可以用vcf2maf工具从vcf转化为MAF格式,至于为什么我想说要转为MAF格式呢,因为有个maftools这个R包,其基础MAF格式可以做很多分析以及可视化工作,特别方便!
maftools手册中提到,如果是ANNOVAR注释结果的文件,可以用其
annovarToMaf将annovar outputs 转化为MAF文件
如果你是其他公共数据,则可以通过人工处理将必要数据整理成MAF格式的文件,maftools需要的必要列有:Hugo_Symbol, Chromosome, Start_Position, End_Position, Reference_Allele, Tumor_Seq_Allele2, Variant_Classification, Variant_Type and Tumor_Sample_Barcode,另外还可以额外再加上VAF (Variant Allele Frequecy)列和amino acid change information
我们可以先用maftools包的测试数据先尝试一遍这个R包的关键几个用法(比较常见的),如:
laml.maf = system.file('extdata', 'tcga_laml.maf.gz', package = 'maftools') #path to TCGA LAML MAF file
laml.clin = system.file('extdata', 'tcga_laml_annot.tsv', package = 'maftools') # clinical information containing survival information and histology. This is optional
laml = read.maf(maf = laml.maf, clinicalData = laml.clin)然后看下laml这个对象存储了哪个内容:
getSampleSummary(laml)
getGeneSummary(laml)
getClinicalData(laml)
getFields(laml)接下来可以看看maftools根据所提供的MAF文件能做哪些分析以及可视化工作了,先是一个summary整合图
plotmafSummary(maf = laml, rmOutlier = TRUE, addStat = 'median', dashboard = TRUE, titvRaw = FALSE)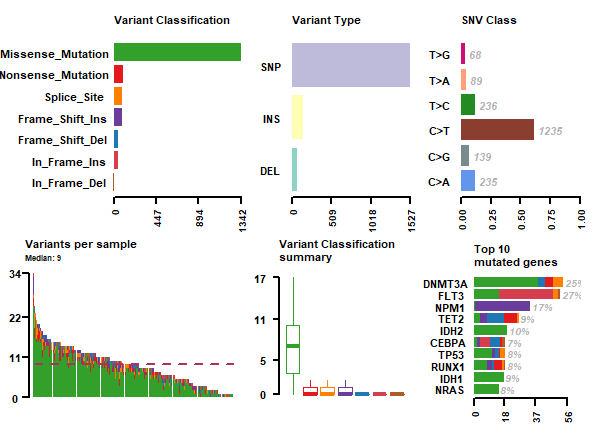
接下来是可能是最为常见的oncoplots图,其oncoplot函数主要是在ComplexHeatmap包的OncoPrint函数基础上做了一定的修改,当然图形展现形式还是一样的,主要展示每个样本在一些基因上突变情况,可以简单的看一下
oncoplot(maf = laml, top = 10, fontSize = 12)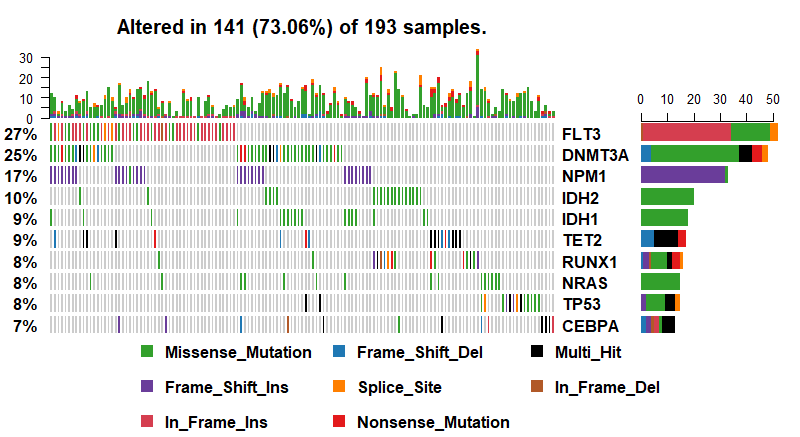
如果你的MAF文件有VAF信息的话,可以简单画个箱线图看看所有样本在不同gene上的VAF分布,vafCol就是指定MAF列中的VAF的列名
plotVaf(maf = laml, vafCol = 'i_TumorVAF_WU')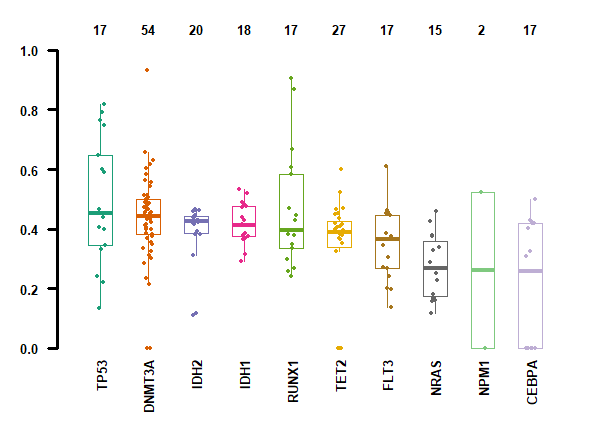
接下来则是我觉得比较有意思的:Detecting cancer driver genes based on positional clustering,这个方法是通过突变位点的位置信息,基于oncodriveCLUST算法来识别cancer genes
laml.sig = oncodrive(maf = laml, AACol = 'Protein_Change', minMut = 5, pvalMethod = 'zscore')
head(laml.sig)
plotOncodrive(res = laml.sig, fdrCutOff = 0.1, useFraction = TRUE)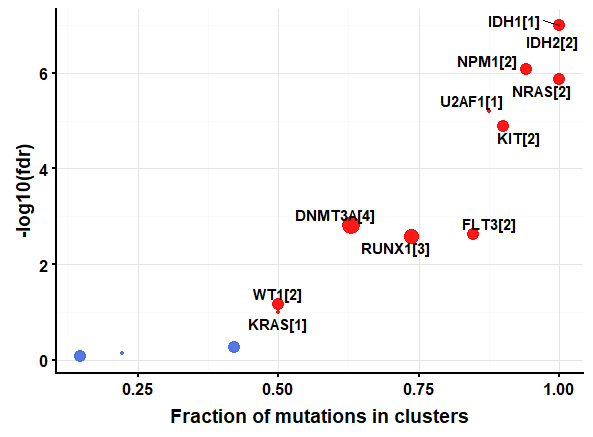
从上图可看出,纵坐标越大表示FDR越小可行度越高;而横坐标在表示那些突变位点聚到一个cluster的比例,而点的大小(中括号中的数字)则表示cluster的数目,所以 IDH1 has a single cluster and all of the 18 mutations are accumulated within that cluster
最后则是这篇笔记的重点内容了,之前有篇博文Mutant-allele tumor heterogeneity(MATH)提到过MATH这个生物标志物,一些文献也对其在不同肿瘤中的预测效果进行了研究,刚好生信技能树的一篇软文使用Mutant-allele tumor heterogeneity(MATH)算法评估肿瘤异质性中提到使用maftools可以计算MATH
maftools的inferHeterogeneity函数可以对MAF格式的数据中的VAF列进行计算MATH,其算法跟传统的MATH不完全一样,其还加入了聚类算法(using mclust),相当于除去了一些outline的VAF值后再计算MATH,用测试数据的话方法如下:
tcga.ab.2972.het = inferHeterogeneity(maf = laml, tsb = 'TCGA-AB-2972', vafCol = 'i_TumorVAF_WU')
> unique(tcga.ab.2972.het$clusterData$MATH)
[1] 11.62157我们也可以拿TCGA的MAF突变数据尝试下,先从TCGA官网上下载相关数据,比如我选择BRCA-mutect2的somatic数据TCGA.BRCA.mutect.somatic.maf(名字原来不是这个,我改了下)
然后就是用maftools导入R中,看下TCGA的MAF文件有多少(很多列。。)
data <- read.maf(maf = "TCGA.BRCA.mutect.somatic.maf")
getFields(data)接着因为要计算MATH,所以还需要先计算一个VAF值(t_alt_count/t_depth)
data@data$VAF <- data@data$t_alt_count/data@data$t_depth然后挑一个样本计算下MATH(这里只用默认参数,具体分析可能会再调试下)
> head(data@data$Tumor_Sample_Barcode)
[1] TCGA-D8-A1XY-01A-11D-A14K-09 TCGA-D8-A1XY-01A-11D-A14K-09
[3] TCGA-D8-A1XY-01A-11D-A14K-09 TCGA-D8-A1XY-01A-11D-A14K-09
[5] TCGA-D8-A1XY-01A-11D-A14K-09 TCGA-D8-A1XY-01A-11D-A14K-09
986 Levels: TCGA-3C-AAAU-01A-11D-A41F-09 ... TCGA-Z7-A8R6-01A-11D-A41F-09
res <- inferHeterogeneity(maf = data, tsb = 'TCGA-D8-A1XY-01A-11D-A14K-09', vafCol = 'VAF')
> unique(res$clusterData$MATH)
[1] 57.76873暂时还没比较过传统方法计算的MATH值和maftools包计算的MATH有多少差异。。但是从思路上来看maftools加上聚类的算法去除outline的方法感觉更加合适点?
参考资料:
What does this following maf column mean? http://bioconductor.org/packages/release/bioc/vignettes/maftools/inst/doc/maftools.html
本文出自于http://www.bioinfo-scrounger.com转载请注明出处